
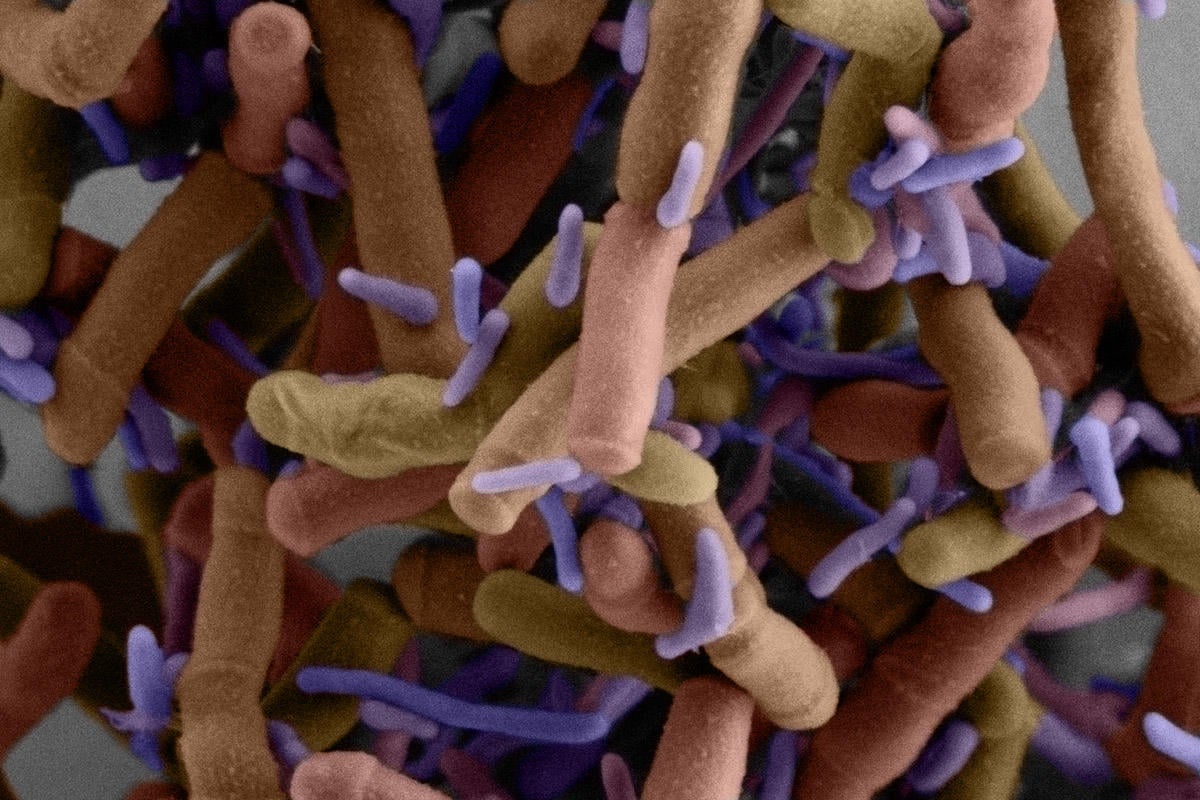
Une micrographie électronique à balayage montre des cellules de minuscules Patskibactéries violettes se développant à la surface de cellules beaucoup plus grandes. Une nouvelle recherche menée par le laboratoire de Joseph Mogos à l’Université de médecine du Wisconsin à Seattle révèle leur cycle de vie, leurs gènes et certains des mécanismes moléculaires qui pourraient être à l’origine de leur mode de vie inhabituel. Cette bactérie épibiotique est Southlakia epibionticum. Crédit image : Yaxi Wang, Wei-Pang Chan et Scott Braswell/Université de Washington
Les scientifiques ont découvert des gènes essentiels au mode de vie inhabituel de minuscules bactéries qui vivent à la surface de bactéries plus grosses.
Les batsibactéries sont un groupe mystérieux de minuscules microbes dotés de méthodes de survie insaisissables. Même si les scientifiques ne peuvent cultiver que quelques-unes de ces espèces, elles font partie d’une famille diversifiée que l’on retrouve dans de nombreux environnements.
Les quelques types de patescibactéries que les chercheurs peuvent cultiver en laboratoire résident à la surface des cellules d’un microbe hôte plus gros. Les patescibactéries ne possèdent généralement pas les gènes nécessaires pour synthétiser de nombreuses molécules essentielles à la vie, par ex. Acides aminés Qui composent les protéines, les acides gras qui composent les membranes et les nucléotides qu’elles contiennent ADN. Cela a conduit les chercheurs à supposer que nombre d’entre eux dépendent d’autres bactéries pour se développer.
Dans une étude récemment publiée dans celluleLes chercheurs donnent un premier aperçu des mécanismes moléculaires à l’origine du mode de vie inhabituel des Patescibactéries. Cette percée a été rendue possible par la découverte d’un moyen de manipuler génétiquement ces bactéries, une avancée qui a ouvert un monde de nouvelles orientations de recherche potentielles.
Nitin S. a déclaré : « Bien que la métagénomique puisse nous renseigner sur les microbes qui vivent sur et dans notre corps, le séquençage de l’ADN à lui seul ne nous donne pas d’informations sur leurs activités bénéfiques ou nocives, en particulier pour les organismes qui n’ont jamais été décrits auparavant », a déclaré Balija de l’institut. de biologie des systèmes à Seattle, qui a contribué à de nombreuses analyses informatiques et systémiques pour l’étude.

Larry A., chercheur sur les bactéries épibiotiques. Gallagher devant un microscope dans le laboratoire de microbiologie de la faculté de médecine de l’Université de Washington. Crédits. Brooke Peterson / Université de Washington
« La capacité de perturber génétiquement les Patescibactéries ouvre la possibilité d’appliquer une puissante analyse systémique pour caractériser rapidement la biologie unique des organismes obligatoires », a-t-il ajouté, faisant référence aux organismes qui doivent subsister sur un autre organisme pour survivre.
L’équipe qui a mené l’étude était dirigée par le laboratoire de Joseph Mougus du Département de microbiologie de l’université. Université de Washington L’École de médecine et le Howard Hughes Medical Institute se sont intéressés à B. batischebacteria pour plusieurs raisons.
Elles font partie des nombreuses bactéries mal comprises dont les séquences d’ADN apparaissent dans les analyses génétiques à grande échelle des génomes de Classer– Riches communautés microbiennes provenant de sources environnementales. Ce matériel génétique est appelé « matière noire microbienne » car on sait peu de choses sur les fonctions qu’il code.
La matière noire microbienne contient probablement des informations sur les voies biochimiques ayant des applications biotechnologiques potentielles, rapportent les chercheurs. cellule papier. Ils contiennent également des preuves des activités moléculaires qui soutiennent un écosystème microbien, ainsi que de la biologie cellulaire des diverses espèces microbiennes rassemblées dans cet écosystème.
Le groupe de Patescibactéries analysé dans cette dernière recherche appartient aux Saccharibactéries. Ces créatures vivent dans divers environnements terrestres et aquatiques, mais sont surtout connues pour habiter la bouche humaine. Ils font partie du microbiome buccal humain au moins depuis l’ère mésolithique et sont liés à la santé bucco-dentaire humaine.
Dans la bouche humaine, la bactérie Saccharomyces a besoin de la compagnie des actinomycètes, qui lui servent d’hôte. Pour mieux comprendre les mécanismes utilisés par la bactérie Saccharomyces pour communiquer avec ses hôtes, les chercheurs ont utilisé la manipulation génétique pour identifier tous les gènes essentiels à la croissance de la bactérie Saccharomyces.

Yaxi Wang, chercheur en bactéries abiotiques, à un poste de travail anaérobie du laboratoire de microbiologie de la faculté de médecine de l’Université de Washington à Seattle. Crédits. Brooke Peterson / Université de Washington
« Nous sommes très heureux d’avoir ce premier aperçu des fonctions génétiques inhabituelles que ces bactéries abritent », a déclaré Mogos, professeur de microbiologie. « En concentrant nos futures études sur ces gènes, nous espérons percer le mystère de la manière dont les glycobactéries exploitent les bactéries hôtes pour leur croissance. »
Les facteurs potentiels d’interaction avec l’hôte révélés dans l’étude comprennent les structures de la surface cellulaire qui peuvent aider les glycobactéries à s’attacher aux cellules hôtes et un système de sécrétion spécialisé qui peut être utilisé pour transporter les nutriments.
Une autre application des travaux des auteurs était la génération de cellules saccharobactériennes exprimant des protéines fluorescentes. À l’aide de ces cellules, les chercheurs ont réalisé une imagerie par microscopie fluorescente accélérée de saccharobactéries se développant avec leurs bactéries hôtes.
S. a noté. « L’imagerie accélérée de cultures de cellules hôtes de S. glycobactéries a révélé la complexité surprenante du cycle de vie de cette bactérie inhabituelle », a déclaré Brooke Peterson, scientifique principale du laboratoire Mogus.
Certaines bactéries Saccharomyces agissent comme des cellules mères en s’attachant à la cellule hôte et en bourgeonnant à plusieurs reprises pour générer une petite progéniture en essaim, rapportent les chercheurs. Ces petits partent à la recherche de nouvelles cellules hôtes. Certains descendants sont à leur tour devenus des cellules mères, tandis que d’autres semblaient interagir de manière improductive avec l’hôte.
Les chercheurs pensent que des études supplémentaires sur la manipulation génétique ouvriront la porte à une compréhension plus large des rôles de ce qu’ils décrivent comme « les riches réserves de matière noire microbienne que contiennent ces organismes » et révéleront peut-être des mécanismes biologiques qui n’ont pas encore été envisagés.
Référence : « La manipulation génétique des Patescibactéries fournit des informations mécanistes sur la matière noire microbienne et le mode de vie abiotique » par Yaxi Wang, Larry A. Gallagher, Pia A. Andrade, Andi Liu, Ian R. Humphreys, Serdar Turkarslan, Kevin J. Cutler, Mario. L. Arrieta-Ortiz, Yaqiao Li, Matthew C. Radey, Jeffrey S. McLean, Qian Cong, David Baker, Nitin S. Baliga, S. Brook Peterson et Joseph D. Mougous, 7 septembre 2023, cellule.
est ce que je: 10.1016/j.cell.2023.08.017
Cette étude interdisciplinaire et collaborative est renforcée par le nouveau projet Centre des interactions microbiennes et du microbiome (en abrégé mim_c), réalisé par Mojos. La mission de mim_c est de réduire les obstacles aux études de recherche sur le microbiome et de favoriser la collaboration en se connectant avec des chercheurs partageant les mêmes idées et issus de diverses disciplines. Ici, mim_c a été le catalyseur qui a rejoint le laboratoire de Mouugous avec l’expert en microbiome oral Jeffrey McLean du département de parodontologie de l’école de médecine dentaire de l’Université du Wisconsin.
Les auteurs principaux de cette étude sont Yaxi Wang et Larry A. Gallagher du département de microbiologie de l’Université du Wisconsin. Les auteurs principaux sont Baliga, Peterson et Mogus. Les biochimistes Qian Kong de l’Université du Texas Sud-Ouest, David Becker et d’autres chercheurs de l’Institut médical de l’Université du Wisconsin ont également contribué à la conception de la protéine, aux côtés de McLean.
Mogus et Baker sont enquêteurs au Howard Hughes Medical Institute. Mogus est titulaire de la chaire Lynn M. et Michael D. Garvey à l’Université de Washington.
L’étude a été financée par des subventions de Instituts nationaux de la santéLa National Science Foundation, l’Agence de réduction des menaces de défense du ministère de la Défense, la Fondation Bill & Melinda Gates et la Fondation Welch.






More Stories
Compenser le sommeil le week-end pourrait réduire d’un cinquième le risque de maladie cardiaque – étude | Maladie cardiaque
Une vache marine préhistorique a été mangée par un crocodile et un requin, selon des fossiles
Un cas de varicelle a été signalé à la prison du comté de Las Vegas